Partie 5 Manipulation de données▲
Cette partie est un peu aride et pas forcément très intuitive. Elle aborde cependant la base de tous les traitements et manipulations de données sous R, et mérite donc que l'on s'y arrête un moment, ou que l'on y revienne un peu plus tard en cas de saturation…
Variables▲
Le type d'objet utilisé par R pour stocker des tableaux de données s'appelle un data frame. Celui-ci comporte des observations en ligne et des variables en colonnes. On accède aux variables d'un data frame avec l'opérateur $.
Dans ce qui suit, on travaillera sur le jeu de données tiré de l'enquête Histoire de vie, fourni avec l'extension rgrs et décrit dans l'annexe B.3.3B.3.3 Le jeu de données hdv2003.
R> library(rgrs)
R> data(hdv2003)
R> d <- hdv2003Mais aussi sur le jeu de données tiré du recensement 1999, décrit dans l'annexe B.3.4B.3.4 Le jeu de données rp99 :
R> data(rp99)Types de variables▲
On peut considérer qu'il existe quatre types de variables dans R :
- les variables numériques, ou quantitatives ;
- les facteurs, qui prennent leurs valeurs dans un ensemble défini de modalités. Elles correspondent en général aux questions fermées d'un questionnaire ;
- les variables caractères, qui contiennent des chaînes de caractères plus ou moins longues. On les utilise pour les questions ouvertes ou les champs libres ;
- les variables booléennes, qui ne peuvent prendre que la valeur vrai (
TRUE) ou faux (FALSE). On les utilise dans R pour les calculs et les recodages.
Pour connaître le type d'une variable donnée, on peut utiliser la fonction class.
|
Résultat de class |
Type de variable |
|---|---|
|
factor |
Facteur |
|
integer |
Numérique |
|
double |
Numérique |
|
numeric |
Numérique |
|
character |
Caractères |
|
logical |
Booléenne |
2.
3.
4.
5.
6.
R> class(d$age)
[1] "integer"
R> class(d$sexe)
[1] "factor"
R> class(c(TRUE, TRUE, FALSE))
[1] "logical"
La fonction str permet également d'avoir un listing de toutes les variables d'un tableau de données et indique le type de chacune d'elle.
Renommer des variables▲
Une opération courante lorsque l'on a importé des variables depuis une source de données externe consiste à renommer les variables importées. Sous R les noms de variables doivent être à la fois courts et explicites tout en obéissant à certaines règles décrites dans la seconde remarque de la section 2.2.1Objets simples.
On peut lister les noms des variables d'un data frame à l'aide de la fonction names :
2.
3.
4.
5.
6.
R> names(d)
[1] "id" "age" "sexe" "nivetud"
[5] "poids" "occup" "qualif" "freres.soeurs"
[9] "clso" "relig" "trav.imp" "trav.satisf"
[13] "hard.rock" "lecture.bd" "peche.chasse" "cuisine"
[17] "bricol" "cinema" "sport" "heures.tv"
Cette fonction peut également être utilisée pour renommer l'ensemble des variables. Si, par exemple on souhaitait passer les noms de toutes les variables en majuscules, on pourrait faire :
2.
3.
4.
5.
6.
7.
8.
9.
R> d.maj <- d
R> names(d.maj) <- c("ID", "AGE", "SEXE", "NIVETUD", "POIDS",
+ "OCCUP", "QUALIF", "FRERES.SOEURS", "CLSO", "RELIG",
+ "TRAV.IMP", "TRAV.SATISF", "HARD.ROCK", "LECTURE.BD",
+ "PECHE.CHASSE", "CUISINE", "BRICOL", "CINEMA", "SPORT",
+ "HEURES.TV")
R> summary(d.maj$SEXE)
Homme Femme
899 1101
Ce type de renommage peut être utile lorsque l'on souhaite passer en revue tous les noms de variables d'un fichier importé, pour les corriger le cas échéant. Pour faciliter un peu ce travail pas forcément passionnant, on peut utiliser la fonction dput :
2.
3.
4.
5.
R> dput(names(d))
c("id", "age", "sexe", "nivetud", "poids", "occup", "qualif",
"freres.soeurs", "clso", "relig", "trav.imp", "trav.satisf",
"hard.rock", "lecture.bd", "peche.chasse", "cuisine", "bricol",
"cinema", "sport", "heures.tv")
On obtient en résultat la liste des variables sous forme de vecteur déclaré. On n'a plus alors qu'à copier/coller cette chaîne, rajouter names(d) <- devant, et modifier un à un les noms des variables.
Si on souhaite seulement modifier le nom d'une variable, on peut utiliser la fonction renomme.variable de l'extension rgrs. Celle-ci prend en argument le tableau de données, le nom actuel de la variable et le nouveau nom. Par exemple, si on veut renommer la variable bricol du tableau de données d en bricolage :
2.
3.
4.
R> d <- renomme.variable(d, "bricol", "bricolage")
R> table(d$bricolage)
Non Oui
1147 853
Facteurs▲
Parmi les différents types de variables, les facteurs (factor) sont à la fois à part et très utilisés, car ils vont correspondre à la plupart des variables issues d'une question fermée dans un questionnaire.
Les facteurs prennent leurs valeurs dans un ensemble de modalités prédéfinies, et ne peuvent en prendre d'autres. La liste des valeurs possibles est donnée par la fonction levels :
2.
R> levels(d$sexe)
[1] "Homme" "Femme"
Si on veut modifier la valeur du sexe du premier individu de notre tableau de données avec une valeur différente, on obtient un message d'erreur et une valeur manquante est utilisée à la place :
2.
3.
4.
R> d$sexe[1] <- "Chihuahua"
R> d$sexe[1]
[1] <NA>
Levels: Homme Femme
On peut très facilement créer un facteur à partir d'une variable de type caractères avec la commande factor :
2.
3.
4.
R> v <- factor(c("H", "H", "F", "H"))
R> v
[1] H H F H
Levels: F H
Par défaut, les niveaux d'un facteur nouvellement créés sont l'ensemble des valeurs de la variable caractères, ordonnées par ordre alphabétique. Cet ordre des niveaux est utilisé à chaque fois que l'on emploie des fonctions comme table, par exemple :
2.
3.
4.
R> table(v)
v
F H
1 3
On peut modifier cet ordre au moment de la création du facteur en utilisant l'option levels :
2.
3.
4.
5.
R> v <- factor(c("H", "H", "F", "H"), levels = c("H", "F"))
R> table(v)
v
H F
3 1
On peut aussi modifier l'ordre des niveaux d'une variable déjà existante :
2.
3.
4.
5.
6.
7.
8.
9.
10.
R> d$qualif <- factor(d$qualif, levels = c("Ouvrier specialise",
+ "Ouvrier qualifie", "Employe", "Technicien", "Profession intermediaire",
+ "Cadre", "Autre"))
R> table(d$qualif)
Ouvrier specialise Ouvrier qualifie Employe
203 292 594
Technicien Profession intermediaire Cadre
86 160 260
Autre
58
Par défaut, les valeurs manquantes ne sont pas considérées comme un niveau de facteur. On peut cependant les transformer en niveau en utilisant l'option exclude=NULL. Ceci signifie cependant qu'elles ne seront plus considérées comme manquantes par R :
2.
3.
4.
5.
6.
R> summary(d$trav.satisf)
Satisfaction Insatisfaction Equilibre NA's
480 117 451 952
R> summary(factor(d$trav.satisf, exclude = NULL))
Satisfaction Insatisfaction Equilibre <NA>
480 117 451 952
Indexation▲
L'indexation est l'une des fonctionnalités les plus puissantes, mais aussi les plus difficiles à maîtriser de R. Il s'agit d'opérations permettant de sélectionner des sous-ensembles d'observations et/ou de variables en fonction de différents critères. L'indexation peut porter sur des vecteurs, des matrices ou des tableaux de données.
Le principe est toujours le même : on indique, entre crochets et à la suite du nom de l'objet à indexer, une série de conditions indiquant ce que l'on garde ou non. Ces conditions peuvent être de différents types.
Indexation directe▲
Le mode le plus simple d'indexation consiste à indiquer la position des éléments à conserver. Dans le cas d'un vecteur cela permet de sélectionner un ou plusieurs éléments de ce vecteur.
Soit le vecteur suivant :
R> v <- c("a", "b", "c", "d", "e", "f", "g")Si on souhaite le premier élément du vecteur, on peut faire :
R> v[1]
[1] "a"Si on souhaite les trois premiers éléments ou les éléments 2, 6 et 7 :
2.
3.
4.
R> v[1:3]
[1] "a" "b" "c"
R> v[c(2, 6, 7)]
[1] "b" "f" "g"
Si on veut le dernier élément :
R> v[length(v)]
[1] "g"Dans le cas de matrices ou de tableaux de données, l'indexation prend deux arguments séparés par une virgule : le premier concerne les lignes et le second les colonnes. Ainsi, si on veut l'élément correspondant à la troisième ligne et à la cinquième colonne du tableau de données d :
R> d[3, 5]
[1] 3994.102On peut également indiquer des vecteurs :
2.
3.
4.
5.
R> d[1:3, 1:2]
id age
1 1 28
2 2 23
3 3 59
Si on laisse l'un des deux critères vide, on sélectionne l'intégralité des lignes ou des colonnes. Ainsi si l'on veut seulement la cinquième colonne ou les deux premières lignes :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
R> d[, 5]
[1] 2634.3982 9738.3958 3994.1025 5731.6615 4329.0940 8674.6994
[7] 6165.8035 12891.6408 7808.8721 2277.1605 704.3227 6697.8682
[13] 7118.4659 586.7714 11042.0774 9958.2287 4836.1393 1551.4846
[19] 3141.1572 27195.8378 14647.9983 8128.0603 1281.9156 11663.3383
[25] 8780.2614 1700.8437 6662.8375 3359.4690 8536.1101 10620.5259
[31] 5264.2953 14161.7597 1339.6196 9243.9153 4512.2959 7871.6452
[37] 1356.9621 7626.3300 1630.2746 2196.2485 5605.9846 8841.2960
[43] 9113.5378 2267.5912 7706.2944 2446.5111 8118.2639 10751.5037
[49] 831.8599 6591.6440 1936.8826 834.3845 3432.5286 11354.8932
[55] 9292.9762 6344.1227 4899.9404 4766.8652 3462.8121 23732.4853
[61] 833.8428 8529.4403 3190.3680 2423.1052 5945.9929 14991.8652
[67] 2062.1124 5702.0623 20604.2642 2634.4861
[ reached getOption("max.print") -- omitted 1930 entries ]]
R> d[1:2, ]
id age sexe nivetud
1 1 28 Femme Enseignement superieur y compris technique superieur
2 2 23 Femme <NA>
poids occup qualif freres.soeurs clso
1 2634.398 Exerce une profession Employe 8 Oui
2 9738.396 Etudiant, eleve <NA> 2 Oui
relig trav.imp trav.satisf hard.rock
1 Ni croyance ni appartenance Peu important Insatisfaction Non
2 Ni croyance ni appartenance <NA> <NA> Non
lecture.bd peche.chasse cuisine bricol cinema sport heures.tv
1 Non Non Oui Non Non Non 0
2 Non Non Non Non Oui Oui 1
Enfin, si on préfixe les arguments avec le signe « - », ceci signifie « tous les éléments sauf ceux indiqués ». Si, par exemple on veut tous les éléments de v sauf le premier :
R> v[-1]
[1] "b" "c" "d" "e" "f" "g"Bien sûr, tous ces critères se combinent et on peut stocker le résultat dans un nouvel objet. Dans cet exemple d2 contiendra les trois premières lignes de d,mais sans les colonnes 2, 6 et 8.
R> d2 <- d[1:3, -c(2, 6, 8)]Indexation par nom▲
Un autre mode d'indexation consiste à fournir non pas un numéro, mais un nom sous forme de chaîne de caractères. On l'utilise couramment pour sélectionner les variables d'un tableau de données. Ainsi, les deux fonctions suivantes sont équivalentes :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
27.
28.
29.
30.
31.
32.
33.
34.
R> d$clso
[1] Oui Oui Non Non Oui
[6] Non Oui Non Oui Non
[11] Oui Oui Oui Oui Oui
[16] Non Non Non Non Non
[21] Oui Oui Non Non Non
[26] Oui Non Non Non Oui
[31] Non Oui Oui Non Non
[36] Oui Oui Non Non Oui
[41] Non Non Oui Non Non
[46] Non Non Oui Oui Non
[51] Non Non Oui Non Oui
[56] Oui Non Non Oui Non
[61] Non Oui Oui Oui Oui
[66] Non Oui Non Non Ne sait pas
[ reached getOption("max.print") -- omitted 1930 entries ]]
Levels: Oui Non Ne sait pas
R> d[, "clso"]
[1] Oui Oui Non Non Oui
[6] Non Oui Non Oui Non
[11] Oui Oui Oui Oui Oui
[16] Non Non Non Non Non
[21] Oui Oui Non Non Non
[26] Oui Non Non Non Oui
[31] Non Oui Oui Non Non
[36] Oui Oui Non Non Oui
[41] Non Non Oui Non Non
[46] Non Non Oui Oui Non
[51] Non Non Oui Non Oui
[56] Oui Non Non Oui Non
[61] Non Oui Oui Oui Oui
[66] Non Oui Non Non Ne sait pas
[ reached getOption("max.print") -- omitted 1930 entries ]]
Levels: Oui Non Ne sait pas
Là aussi on peut utiliser un vecteur pour sélectionner plusieurs noms et récupérer un « sous-tableau » de données :
R> d2 <- d[, c("id", "sexe", "age")]Les noms peuvent également être utilisés pour les observations (lignes) d'un tableau de données si celles-ci ont été munies d'un nom avec la fonction row.names. Par défaut les noms de ligne sont leur numéro d'ordre, mais on peut leur assigner comme nom la valeur d'une variable d'identifiant. Ainsi, on peut assigner aux lignes du jeu de données rp99 le nom des communes correspondantes :
R> row.names(rp99) <- rp99$nomOn peut alors accéder directement aux communes en donnant leur nom :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
R> rp99[c("VILLEURBANNE", "OULLINS"), ]
nom code pop.act pop.tot pop15 nb.rp agric
VILLEURBANNE VILLEURBANNE 69266 57252 124152 103157 55136 0.02095997
OULLINS OULLINS 69149 11849 25186 20880 11091 0.10127437
artis cadres interm empl ouvr retr
VILLEURBANNE 5.143925 13.13841 25.72312 31.41550 23.07343 36.65374
OULLINS 4.818972 10.20339 27.42847 31.53009 24.37336 41.54781
tx.chom etud dipl.sup dipl.aucun proprio hlm
VILLEURBANNE 14.82394 15.50646 9.744370 16.90045 37.61970 23.33684
OULLINS 10.64225 10.62739 7.624521 14.31513 51.51023 14.56136
locataire maison
VILLEURBANNE 32.76988 6.532937
OULLINS 29.91615 17.708052
Par contre, il n'est pas possible d'utiliser directement l'opérateur « - » comme pour l'indexation directe. On doit effectuer une pirouette un peu compliquée utilisant la fonction which. Celle-ci renvoie les positions des éléments satisfaisant une condition. On peut ainsi faire :
2.
3.
R> which(names(d) == "qualif")
[1] 7
R> d2 <- d[, -which(names(d) == "qualif")]
pour sélectionner toutes les colonnes sauf celle qui s'appelle qualif.
Indexation par conditions▲
Tests et conditions
Une condition est une expression logique dont le résultat est soit TRUE (vrai) soit FALSE (faux).
Une condition comprend la plupart du temps un opérateur de comparaison. Les plus courants sont les suivants :
|
Opérateur |
Signification |
|---|---|
|
égal à |
|
|
différent de |
|
|
strictement supérieur à |
|
|
strictement inférieur à |
|
|
supérieur ou égal à |
|
|
inférieur ou égal à |
Voyons tout de suite un exemple :
2.
3.
4.
5.
6.
7.
8.
9.
R> d$sexe == "Homme"
[1] FALSE FALSE TRUE TRUE FALSE FALSE FALSE TRUE FALSE TRUE FALSE
[12] TRUE FALSE FALSE FALSE FALSE TRUE FALSE TRUE FALSE FALSE TRUE
[23] FALSE FALSE FALSE TRUE FALSE TRUE TRUE TRUE TRUE TRUE TRUE
[34] TRUE FALSE FALSE TRUE FALSE FALSE TRUE FALSE TRUE TRUE FALSE
[45] FALSE TRUE FALSE FALSE FALSE FALSE TRUE FALSE TRUE FALSE TRUE
[56] FALSE FALSE FALSE TRUE FALSE FALSE TRUE TRUE TRUE TRUE FALSE
[67] TRUE TRUE FALSE FALSE
[ reached getOption("max.print") -- omitted 1930 entries ]]
Que s'est-il passé ? Nous avons fourni à R une condition qui signifie « la valeur de la variable sexe vaut "Homme" ». Et il nous a renvoyé un vecteur avec autant d'éléments qu'il y a d'observations dans d, et dont la valeur est TRUE si l'observation correspond à un homme, et FALSE dans les autres cas.
Prenons un autre exemple. On n'affichera cette fois que les premiers éléments de notre variable d'intérêt à l'aide de la fonction head :
2.
3.
4.
R> head(d$age)
[1] 28 23 59 34 71 35
R> head(d$age > 40)
[1] FALSE FALSE TRUE FALSE TRUE FALSE
On voit bien ici qu'à chaque élément du vecteur d$age dont la valeur est supérieure à 40 correspond un élément TRUE dans le résultat de la condition.
On peut combiner ou modifier des conditions à l'aide des opérateurs logiques habituels :
Comment les utilise-t-on ? Voyons tout de suite des exemples. Supposons que je veuille déterminer quels sont dans mon échantillon les hommes ouvriers spécialisés :
2.
3.
4.
5.
6.
7.
8.
9.
R> d$sexe == "Homme" & d$qualif == "Ouvrier specialise"
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[12] FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE
[23] FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE FALSE NA
[34] NA FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[45] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[56] FALSE FALSE FALSE FALSE FALSE FALSE NA FALSE NA FALSE FALSE
[67] FALSE FALSE FALSE FALSE
[ reached getOption("max.print") -- omitted 1930 entries ]]
Si je souhaite identifier les personnes qui bricolent ou qui font la cuisine :
2.
3.
4.
5.
6.
7.
8.
9.
R> d$bricol == "Oui" | d$cuisine == "Oui"
[1] TRUE FALSE FALSE TRUE FALSE FALSE TRUE TRUE FALSE FALSE TRUE
[12] TRUE TRUE TRUE TRUE FALSE TRUE TRUE FALSE FALSE TRUE FALSE
[23] TRUE FALSE TRUE FALSE TRUE FALSE TRUE TRUE TRUE TRUE TRUE
[34] TRUE TRUE FALSE TRUE FALSE FALSE TRUE FALSE FALSE TRUE FALSE
[45] FALSE TRUE TRUE TRUE FALSE FALSE TRUE TRUE FALSE FALSE FALSE
[56] TRUE FALSE TRUE FALSE TRUE TRUE TRUE TRUE TRUE FALSE TRUE
[67] TRUE TRUE TRUE TRUE
[ reached getOption("max.print") -- omitted 1930 entries ]]
Si je souhaite isoler les femmes qui ont entre 20 et 34 ans :
2.
3.
4.
5.
6.
7.
8.
9.
R> d$sexe == "Femme" & d$age >= 20 & d$age <= 34
[1] TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE
[12] FALSE FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE TRUE FALSE
[23] FALSE FALSE TRUE FALSE TRUE FALSE FALSE FALSE FALSE FALSE FALSE
[34] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[45] FALSE FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE TRUE FALSE
[56] TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE
[67] FALSE FALSE FALSE FALSE
[ reached getOption("max.print") -- omitted 1930 entries ]]
Si je souhaite récupérer les enquêtés qui ne sont pas cadres, on peut utiliser l'une des deux formes suivantes :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
R> d$qualif != "Cadre"
[1] TRUE NA TRUE TRUE TRUE TRUE TRUE TRUE NA TRUE TRUE
[12] TRUE TRUE NA NA TRUE FALSE NA TRUE TRUE TRUE TRUE
[23] TRUE NA TRUE FALSE TRUE TRUE TRUE TRUE TRUE TRUE NA
[34] NA TRUE TRUE TRUE NA TRUE FALSE TRUE TRUE FALSE FALSE
[45] NA TRUE TRUE NA TRUE FALSE TRUE TRUE TRUE NA TRUE
[56] TRUE NA FALSE TRUE NA TRUE NA TRUE NA TRUE TRUE
[67] TRUE TRUE TRUE NA
[ reached getOption("max.print") -- omitted 1930 entries ]]
R> !(d$qualif == "Cadre")
[1] TRUE NA TRUE TRUE TRUE TRUE TRUE TRUE NA TRUE TRUE
[12] TRUE TRUE NA NA TRUE FALSE NA TRUE TRUE TRUE TRUE
[23] TRUE NA TRUE FALSE TRUE TRUE TRUE TRUE TRUE TRUE NA
[34] NA TRUE TRUE TRUE NA TRUE FALSE TRUE TRUE FALSE FALSE
[45] NA TRUE TRUE NA TRUE FALSE TRUE TRUE TRUE NA TRUE
[56] TRUE NA FALSE TRUE NA TRUE NA TRUE NA TRUE TRUE
[67] TRUE TRUE TRUE NA
[ reached getOption("max.print") -- omitted 1930 entries ]]
Lorsque l'on mélange « et » et « ou » il est nécessaire d'utiliser des parenthèses pour différencier les blocs. La condition suivante identifie les femmes qui sont soit cadres, soit employées :
2.
3.
4.
5.
6.
7.
8.
9.
R> d$sexe == "Femme" & (d$qualif == "Employe" | d$qualif == "Cadre")
[1] TRUE NA FALSE FALSE TRUE TRUE FALSE FALSE NA FALSE TRUE
[12] FALSE TRUE NA NA TRUE FALSE NA FALSE FALSE TRUE FALSE
[23] TRUE NA FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[34] FALSE FALSE TRUE FALSE NA TRUE FALSE TRUE FALSE FALSE TRUE
[45] NA FALSE FALSE NA TRUE TRUE FALSE TRUE FALSE NA FALSE
[56] FALSE NA TRUE FALSE NA TRUE FALSE FALSE FALSE FALSE TRUE
[67] FALSE FALSE TRUE NA
[ reached getOption("max.print") -- omitted 1930 entries ]]
L'opérateur %in% peut être très utile : il teste si une valeur fait partie des éléments d'un vecteur. Ainsi on pourrait remplacer la condition précédente par :
2.
3.
4.
5.
6.
7.
8.
9.
R> d$sexe == "Femme" & d$qualif %in% c("Employe", "Cadre")
[1] TRUE FALSE FALSE FALSE TRUE TRUE FALSE FALSE FALSE FALSE TRUE
[12] FALSE TRUE FALSE FALSE TRUE FALSE FALSE FALSE FALSE TRUE FALSE
[23] TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[34] FALSE FALSE TRUE FALSE FALSE TRUE FALSE TRUE FALSE FALSE TRUE
[45] FALSE FALSE FALSE FALSE TRUE TRUE FALSE TRUE FALSE FALSE FALSE
[56] FALSE FALSE TRUE FALSE FALSE TRUE FALSE FALSE FALSE FALSE TRUE
[67] FALSE FALSE TRUE FALSE
[ reached getOption("max.print") -- omitted 1930 entries ]]
Enfin, signalons que l'on peut utiliser les fonctions table ou summary pour avoir une idée du résultat de notre condition :
2.
3.
4.
5.
6.
7.
8.
9.
R> table(d$sexe)
Homme Femme
899 1101
R> table(d$sexe == "Homme")
FALSE TRUE
1101 899
R> summary(d$sexe == "Homme")
Mode FALSE TRUE NA's
logical 1101 899 0
Utilisation pour l'indexation
L'utilisation des conditions pour l'indexation est assez simple : si on indexe un vecteur avec un vecteur booléen, seuls les éléments correspondant à TRUE seront conservés.
Ainsi, si on fait :
R> dh <- d[d$sexe == "Homme", ]on obtiendra un nouveau tableau de données comportant l'ensemble des variables de d, mais seulement les observations pour lesquelles d$sexe vaut « Homme ».
La plupart du temps ce type d'indexation s'applique aux lignes, mais on peut aussi l'utiliser sur les colonnes d'un tableau de données. L'exemple suivant, un peu compliqué, sélectionne uniquement les variables dont le nom commence par « a » ou « s » :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
25.
26.
R> d[, substr(names(d), 0, 1) %in% c("a", "s")]
age sexe sport
1 28 Femme Non
2 23 Femme Oui
3 59 Homme Oui
4 34 Homme Oui
5 71 Femme Non
6 35 Femme Oui
7 60 Femme Non
8 47 Homme Non
9 20 Femme Non
10 28 Homme Oui
11 65 Femme Non
12 47 Homme Oui
13 63 Femme Non
14 67 Femme Non
15 76 Femme Non
16 49 Femme Non
17 62 Homme Oui
18 20 Femme Oui
19 70 Homme Non
20 39 Femme Oui
21 30 Femme Non
22 30 Homme Non
23 37 Femme Oui
[getOption("max.print") est atteint -- 1977 lignes omises ]]
On peut évidemment combiner les différents types d'indexation. L'exemple suivant sélectionne les femmes de plus de 40 ans et ne conserve que les variables qualif et bricol.
R> d2 <- d[d$sexe == "Femme" & d$age > 40, c("qualif", "bricol")]Valeurs manquantes dans les conditions
Une remarque importante : quand l'un des termes d'une condition comporte une valeur manquante (NA), le résultat de cette condition n'est pas toujours TRUE ou FALSE, il peut aussi être à son tour une valeur manquante.
2.
3.
4.
5.
R> v <- c(1:5, NA)
R> v
[1] 1 2 3 4 5 NA
R> v > 3
[1] FALSE FALSE FALSE TRUE TRUE NA
On voit que le test NA > 3 ne renvoie ni vrai ni faux, mais NA.
Le résultat d'une condition peut donc comporter un grand nombre de valeurs manquantes :
2.
3.
R> summary(d$trav.satisf == "Satisfaction")
Mode FALSE TRUE NA's
logical 568 480 952
Une autre conséquence importante de ce comportement est que l'on ne peut pas utiliser l'opérateur == NA pour tester la présence de valeurs manquantes. On utilisera à la place la fonction ad hoc is.na.
On comprendra mieux le problème avec l'exemple suivant :
2.
3.
4.
5.
6.
7.
R> v <- c(1, NA)
R> v
[1] 1 NA
R> v == NA
[1] NA NA
R> is.na(v)
[1] FALSE TRUE
Pour compliquer encore un peu le tout, lorsque l'on utilise une condition pour l'indexation, si la condition renvoie NA, l'élément est sélectionné, comme s'il valait TRUE. Ceci aura donc des conséquences pour l'extraction de sous-populations, comme indiqué section 5.3.1Par indexation.
Indexation et assignation▲
Dans tous les exemples précédents, on a utilisé l'indexation pour extraire une partie d'un vecteur ou d'un tableau de données, en plaçant l'opération d'indexation à droite de l'opérateur <-.
Mais l'indexation peut également être placée à gauche de cet opérateur. Dans ce cas, les éléments sélectionnés par l'indexation sont alors remplacés par les valeurs indiquées à droite de l'opérateur <-.
Ceci est parfaitement incompréhensible. Prenons donc un exemple simple :
2.
3.
4.
5.
6.
R> v <- 1:5
R> v
[1] 1 2 3 4 5
R> v[1] <- 3
R> v
[1] 3 2 3 4 5
Cette fois, au lieu d'utiliser quelque chose comme x <- v[1], qui aurait placé la valeur du premier élément de v dans x, on a utilisé v[1] <- 3, ce qui a mis à jour le premier élément de v avec la valeur 3.
Ceci fonctionne également pour les tableaux de données et pour les différents types d'indexation évoqués précédemment :
R> d[257, "sexe"] <- "Homme"Enfin on peut modifier plusieurs éléments d'un seul coup soit en fournissant un vecteur, soit en profitant du mécanisme de recyclage. Les deux commandes suivantes sont ainsi rigoureusement équivalentes :
R> d[c(257, 438, 889), "sexe"] <- c("Homme", "Homme", "Homme")
R> d[c(257, 438, 889), "sexe"] <- "Homme"On commence à voir comment l'utilisation de l'indexation par conditions et de l'assignation va nous permettre de faire des recodages.
R> d$age[d$age >= 20 & d$age <= 30] <- "20-30 ans"
R> d$age[is.na(d$age)] <- "Inconnu"Sous-populations▲
Par indexation▲
La première manière de construire des sous-populations est d'utiliser l'indexation par conditions. On peut ainsi facilement sélectionner une partie des observations suivant un ou plusieurs critères et placer le résultat dans un nouveau tableau de données.
Par exemple, si on souhaite isoler les hommes et les femmes :
2.
3.
4.
5.
6.
7.
8.
9.
R> dh <- d[d$sexe == "Homme", ]
R> df <- d[d$sexe == "Femme", ]
R> table(d$sexe)
Homme Femme
899 1101
R> dim(dh)
[1] 899 20
R> dim(df)
[1] 1101 20
On a à partir de là trois tableaux de données, d comportant la population totale, dh seulement les hommes et df seulement les femmes.
On peut évidemment combiner plusieurs critères :
2.
3.
R> dh.25 <- d[d$sexe == "Homme" & d$age <= 25, ]
R> dim(dh.25)
[1] 86 20
Si on utilise directement l'indexation, il convient cependant d'être extrêmement prudent avec les valeurs manquantes. Comme indiqué précédemment, l'existence d'une valeur manquante dans une condition fait que celle-ci est évaluée en NA et finalement sélectionnée par l'indexation :
2.
3.
4.
5.
6.
R> summary(d$trav.satisf)
Satisfaction Insatisfaction Equilibre NA's
480 117 451 952
R> d.satisf <- d[d$trav.satisf == "Satisfaction", ]
R> dim(d.satisf)
[1] 1432 20
Comme on le voit, ici d.satisf contient les individus ayant la modalité Satisfaction, mais aussi ceux ayant une valeur manquante NA. C'est pourquoi il faut toujours soit vérifier au préalable que l'on n'a pas de valeurs manquantes dans les variables de la condition, soit exclure explicitement les NA de la manière suivante :
2.
3.
R> d.satisf <- d[d$trav.satisf == "Satisfaction" & !is.na(d$trav.satisf), ]
R> dim(d.satisf)
[1] 480 20
C'est notamment pour cette raison que l'on préférera le plus souvent utiliser la fonction subset.
Fonction subset▲
La fonction subset permet d'extraire des sous-populations de manière plus simple et un peu plus intuitive que l'indexation directe.
Celle-ci prend trois arguments principaux :
- le nom de l'objet de départ ;
- une condition sur les observations (subset) ;
- éventuellement une condition sur les colonnes (select).
Reprenons tout de suite un exemple déjà vu :
R> dh <- subset(d, sexe == "Homme")
R> df <- subset(d, sexe == "Femme")L'utilisation de subset présente plusieurs avantages. Le premier est d'économiser quelques touches. On n'est en effet pas obligé de saisir le nom du tableau de données dans la condition sur les lignes. Ainsi les deux commandes suivantes sont équivalentes :
R> dh <- subset(d, d$sexe == "Homme")
R> dh <- subset(d, sexe == "Homme")Le second avantage est que subset s'occupe du problème des valeurs manquantes évoquées précédemment et les exclut de lui-même, contrairement au comportement par défaut :
2.
3.
4.
5.
6.
7.
8.
9.
R> summary(d$trav.satisf)
Satisfaction Insatisfaction Equilibre NA's
480 117 451 952
R> d.satisf <- d[d$trav.satisf == "Satisfaction", ]
R> dim(d.satisf)
[1] 1432 20
R> d.satisf <- subset(d, trav.satisf == "Satisfaction")
R> dim(d.satisf)
[1] 480 20
Enfin, l'utilisation de l'argument select est simplifiée pour l'expression de condition sur les colonnes. On peut ainsi spécifier les noms de variables sans guillemets et leur appliquer directement l'opérateur d'exclusion - :
R> d2 <- subset(d, select = c(sexe, sport))
R> d2 <- subset(d, age > 25, select = -c(id, age, bricol))Fonction tapply▲
Cette section documente une fonction qui peut être très utile, mais pas forcément indispensable au départ.
La fonction tapply n'est qu'indirectement liée à la notion de sous-population, mais peut permettre d'éviter d'avoir à créer ces sous-populations dans certains cas.
Son fonctionnement est assez simple, mais pas forcément intuitif. La fonction prend trois arguments : un vecteur, un facteur et une fonction. Elle applique ensuite la fonction aux éléments du vecteur correspondant à un même niveau du facteur. Vite, un exemple !
R> tapply(d$age, d$sexe, mean)
Homme Femme
48.16129 48.15350Qu'est-ce que ça signifie ? Ici tapply a sélectionné toutes les observations correspondant à « Homme », puis appliqué la fonction mean aux valeurs de age correspondantes. Puis elle a fait de même pour les observations correspondant à « Femme ». On a donc ici la moyenne d'âge chez les hommes et chez les femmes.
On peut fournir à peu près n'importe quelle fonction à tapply :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
R> tapply(d$bricol, d$sexe, freq)
$Homme
n %
Non 384 42.7
Oui 515 57.3
NA 0 0.0
$Femme
n %
Non 763 69.3
Oui 338 30.7
NA 0 0.0
Les arguments supplémentaires fournis à tapply sont en fait fournis directement à la fonction appelée.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
R> tapply(d$bricol, d$sexe, freq, total = TRUE)
$Homme
n %
Non 384 42.7
Oui 515 57.3
NA 0 0.0
Total 899 100.0
$Femme
n %
Non 763 69.3
Oui 338 30.7
NA 0 0.0
Total 1101 100.0
Recodages▲
Le recodage de variables est une opération extrêmement fréquente lors du traitement d'enquête. Celui-ci utilise soit l'une des formes d'indexation décrites précédemment, soit des fonctions ad hoc de R.
On passe ici en revue différents types de recodage parmi les plus courants. Les exemples s'appuient, comme précédemment, sur l'extrait de l'enquête Histoire de vie :
R> data(hdv2003)
R> d <- hdv2003Convertir une variable▲
Il peut arriver que l'on veuille transformer une variable d'un type dans un autre.
Par exemple, on peut considérer que la variable numérique freres.soeurs est une « fausse » variable numérique et qu'une représentation sous forme de facteur serait plus adéquate. Dans ce cas il suffit de faire appel à la fonction factor :
2.
3.
4.
R> d$fs.fac <- factor(d$freres.soeurs)
R> levels(d$fs.fac)
[1] "0" "1" "2" "3" "4" "5" "6" "7" "8" "9" "10" "11" "12" "13"
[15] "14" "15" "16" "18" "22"
La conversion d'une variable caractères en facteur se fait de la même manière.
La conversion d'un facteur ou d'une variable numérique en variable caractères peut se faire à l'aide de la fonction as.character :
2.
R> d$fs.char <- as.character(d$freres.soeurs)
R> d$qualif.char <- as.character(d$qualif)
La conversion d'un facteur en caractères est fréquemment utilisée lors des recodages du fait qu'il est impossible d'ajouter de nouvelles modalités à un facteur. Par exemple, la première des commandes suivantes génère un message d'avertissement, tandis que les deux autres fonctionnent :
2.
3.
R> d$qualif[d$qualif == "Ouvrier specialise"] <- "Ouvrier"
R> d$qualif.char <- as.character(d$qualif)
R> d$qualif.char[d$qualif.char == "Ouvrier specialise"] <- "Ouvrier"
Dans le premier cas, le message d'avertissement indique que toutes les modalités « Ouvrier specialise » de notre variable qualif ont été remplacées par des valeurs manquantes NA.
Enfin, une variable de type caractères dont les valeurs seraient des nombres peut être convertie en variable numérique avec la fonction as.numeric. Si on souhaite convertir un facteur en variable numérique, il faut d'abord le convertir en variable de classe caractère :
R> d$fs.num <- as.numeric(as.character(d$fs.fac))Découper une variable numérique en classes▲
Le premier type de recodage consiste à découper une variable de type numérique en un certain nombre de classes. On utilise pour cela la fonction cut.
Celle-ci prend, outre la variable à découper, un certain nombre d'arguments :
- breaks indique soit le nombre de classes souhaité, soit, si on lui fournit un vecteur, les limites des classes ;
- labels permet de modifier les noms de modalités attribués aux classes ;
- include.lowest et right influent sur la manière dont les valeurs situées à la frontière des classes seront incluses ou exclues ;
- dig.lab indique le nombre de chiffres après la virgule à conserver dans les noms de modalités.
Prenons tout de suite un exemple et tentons de découper notre variable age en cinq classes et de placer le résultat dans une nouvelle variable nommée age5cl :
2.
3.
4.
R> d$age5cl <- cut(d$age, 5)
R> table(d$age5cl)
(17.9,33.8] (33.8,49.6] (49.6,65.4] (65.4,81.2] (81.2,97.1]
454 628 556 319 43
Par défaut R nous a bien créé cinq classes d'amplitudes égales. La première classe va de 16,9 à 32,2 ans (en fait de 17 à 32), etc.
Les frontières de classe seraient plus présentables si elles utilisaient des nombres entiers. On va donc spécifier manuellement le découpage souhaité, par tranches de 20 ans :
2.
3.
4.
R> d$age20 <- cut(d$age, c(0, 20, 40, 60, 80, 100))
R> table(d$age20)
(0,20] (20,40] (40,60] (60,80] (80,100]
72 660 780 436 52
On aurait pu tenir compte des âges extrêmes pour la première et la dernière valeur :
2.
3.
4.
5.
6.
R> range(d$age)
[1] 18 97
R> d$age20 <- cut(d$age, c(17, 20, 40, 60, 80, 93))
R> table(d$age20)
(17,20] (20,40] (40,60] (60,80] (80,93]
72 660 780 436 50
Les symboles dans les noms attribués aux classes ont leur importance : ( signifie que la frontière de la classe est exclue, tandis que [ signifie qu'elle est incluse. Ainsi, (20,40] signifie « strictement supérieur à 20 et inférieur ou égal à 40 ».
On remarque que du coup, dans notre exemple précédent, la valeur minimale, 17, est exclue de notre première classe, et qu'une observation est donc absente de ce découpage. Pour résoudre ce problème, on peut soit faire commencer la première classe à 16, soit utiliser l'option include.lowest=TRUE :
2.
3.
4.
5.
6.
7.
8.
R> d$age20 <- cut(d$age, c(16, 20, 40, 60, 80, 93))
R> table(d$age20)
(16,20] (20,40] (40,60] (60,80] (80,93]
72 660 780 436 50
R> d$age20 <- cut(d$age, c(17, 20, 40, 60, 80, 93), include.lowest = TRUE)
R> table(d$age20)
[17,20] (20,40] (40,60] (60,80] (80,93]
72 660 780 436 50
On peut également modifier le sens des intervalles avec l'option right=FALSE, et indiquer manuellement les noms des modalités avec labels :
2.
3.
4.
5.
6.
7.
8.
9.
R> d$age20 <- cut(d$age, c(16, 20, 40, 60, 80, 93), right = FALSE, include.lowest = TRUE)
R> table(d$age20)
[16,20) [20,40) [40,60) [60,80) [80,93]
48 643 793 454 60
R> d$age20 <- cut(d$age, c(17, 20, 40, 60, 80, 93), include.lowest = TRUE,
+ labels = c("<20 ans", "21-40 ans", "41-60 ans", "61-80 ans", ">80 ans"))
R> table(d$age20)
<20 ans 21-40 ans 41-60 ans 61-80 ans >80 ans
72 660 780 436 50
Enfin, l'extension rgrs propose une fonction quant.cut permettant de découper une variable numérique en un nombre de classes donné ayant des effectifs semblables. Il suffit de lui passer le nombre de classes en argument :
2.
3.
4.
R> d$age6cl <- quant.cut(d$age, 6)
R> table(d$age6cl)
[18,30) [30,39) [39,48) [48,55.667) [55.667,66) [66,97]
302 337 350 344 305 362
quant.cut admet les mêmes autres options que cut (include.lowest, right, labels...).
Regrouper les modalités d'une variable▲
Pour regrouper les modalités d'une variable qualitative (d'un facteur le plus souvent), on peut utiliser directement l'indexation.
Ainsi, si on veut recoder la variable qualif dans une variable qualif.reg plus « compacte », on peut utiliser :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
R> table(d$qualif)
Ouvrier specialise, Ouvrier qualifie, Technicien
203, 292, 86
Profession intermediaire, Cadre, Employe
160, 260, 594
Autre
58
R> d$qualif.reg[d$qualif == "Ouvrier specialise"] <- "Ouvrier"
R> d$qualif.reg[d$qualif == "Ouvrier qualifie"] <- "Ouvrier"
R> d$qualif.reg[d$qualif == "Employe"] <- "Employe"
R> d$qualif.reg[d$qualif == "Profession intermediaire"] <- "Intermediaire"
R> d$qualif.reg[d$qualif == "Technicien"] <- "Intermediaire"
R> d$qualif.reg[d$qualif == "Cadre"] <- "Cadre"
R> d$qualif.reg[d$qualif == "Autre"] <- "Autre"
R> table(d$qualif.reg)
Autre Cadre Employe Intermediaire Ouvrier
58 260 594 246 495
On aurait pu représenter ce recodage de manière plus compacte, notamment en commençant par copier le contenu de qualif dans qualif.reg, ce qui permet de ne pas s'occuper de ce qui ne change pas. Il est cependant nécessaire de ne pas copier qualif sous forme de facteur, sinon on ne pourrait ajouter de nouvelles modalités. On copie donc la version caractères de qualif grâce à la fonction as.character :
2.
3.
4.
5.
6.
7.
8.
R> d$qualif.reg <- as.character(d$qualif)
R> d$qualif.reg[d$qualif == "Ouvrier specialise"] <- "Ouvrier"
R> d$qualif.reg[d$qualif == "Ouvrier qualifie"] <- "Ouvrier"
R> d$qualif.reg[d$qualif == "Profession intermediaire"] <- "Intermediaire"
R> d$qualif.reg[d$qualif == "Technicien"] <- "Intermediaire"
R> table(d$qualif.reg)
Autre Cadre Employe Intermediaire Ouvrier
58 260 594 246 495
On peut faire une version encore plus compacte en utilisant l'opérateur logique ou (|) :
2.
3.
4.
5.
6.
R> d$qualif.reg <- as.character(d$qualif)
R> d$qualif.reg[d$qualif == "Ouvrier specialise" | d$qualif == "Ouvrier qualifie"] <- "Ouvrier"
R> d$qualif.reg[d$qualif == "Profession intermediaire" | d$qualif == "Technicien"] <- "Intermediaire"
R> table(d$qualif.reg)
Autre Cadre Employe Intermediaire Ouvrier
58 260 594 246 495
Enfin, pour terminer ce petit tour d'horizon, on peut également remplacer l'opérateur | par %in%, qui peut parfois être plus lisible :
2.
3.
4.
5.
6.
R> d$qualif.reg <- as.character(d$qualif)
R> d$qualif.reg[d$qualif %in% c("Ouvrier specialise", "Ouvrier qualifie")] <- "Ouvrier"
R> d$qualif.reg[d$qualif %in% c("Profession intermediaire", "Technicien")] <- "Intermediaire"
R> table(d$qualif.reg)
Autre Cadre Employe Intermediaire Ouvrier
58 260 594 246 495
Dans tous les cas le résultat obtenu est une variable de type caractère. On pourra la convertir en facteur par un simple :
R> d$qualif.reg <- factor(d$qualif.reg)Si on souhaite recoder les valeurs manquantes, il suffit de faire appel à la fonction is.na :
2.
3.
4.
5.
6.
7.
8.
R> table(d$trav.satisf)
Satisfaction,Insatisfaction, Equilibre
480, 117, 451
R> d$trav.satisf.reg <- as.character(d$trav.satisf)
R> d$trav.satisf.reg[is.na(d$trav.satisf)] <- "Valeur manquante"
R> table(d$trav.satisf.reg)
Equilibre Insatisfaction Satisfaction Valeur manquante
451 117 480 952
Variables calculées▲
La création d'une variable numérique à partir de calculs sur une ou plusieurs autres variables numériques se fait très simplement.
Supposons que l'on souhaite calculer une variable indiquant l'écart entre le nombre d'heures passées à regarder la télévision et la moyenne globale de cette variable. On pourrait alors faire :
2.
3.
4.
5.
6.
7.
8.
9.
R> range(d$heures.tv, na.rm = TRUE)
[1] 0 12
R> mean(d$heures.tv, na.rm = TRUE)
[1] 2.246566
R> d$ecart.heures.tv <- d$heures.tv - mean(d$heures.tv, na.rm = TRUE)
R> range(d$ecart.heures.tv, na.rm = TRUE)
[1] -2.246566 9.753434
R> mean(d$ecart.heures.tv, na.rm = TRUE)
[1] 4.714578e-17
Autre exemple tiré du jeu de données rp99 : si on souhaite calculer le pourcentage d'actifs dans chaque commune, on peut diviser la population active pop.act par la population totale pop.tot.
R> rp99$part.actifs <- rp99$pop.act/rp99$pop.tot * 100Combiner plusieurs variables▲
La combinaison de plusieurs variables se fait à l'aide des techniques d'indexation déjà décrites précédemment. Le plus compliqué est d'arriver à formuler des conditions parfois complexes de manière rigoureuse.
On peut ainsi vouloir combiner plusieurs variables qualitatives en une seule :
2.
3.
4.
5.
6.
7.
8.
9.
10.
R> d$act.manuelles <- NA
R> d$act.manuelles[d$cuisine == "Oui" & d$bricol == "Oui"] <- "Cuisine et Bricolage"
R> d$act.manuelles[d$cuisine == "Oui" & d$bricol == "Non"] <- "Cuisine seulement"
R> d$act.manuelles[d$cuisine == "Non" & d$bricol == "Oui"] <- "Bricolage seulement"
R> d$act.manuelles[d$cuisine == "Non" & d$bricol == "Non"] <- "Ni cuisine ni bricolage"
R> table(d$act.manuelles)
Bricolage seulement Cuisine et Bricolage Cuisine seulement
437 416 465
Ni cuisine ni bricolage
682
On peut également combiner variables qualitatives et variables quantitatives :
2.
3.
4.
5.
6.
7.
8.
9.
10.
R> d$age.sexe <- NA
R> d$age.sexe[d$sexe == "Homme" & d$age < 40] <- "Homme moins de 40 ans"
R> d$age.sexe[d$sexe == "Homme" & d$age >= 40] <- "Homme plus de 40 ans"
R> d$age.sexe[d$sexe == "Femme" & d$age < 40] <- "Femme moins de 40 ans"
R> d$age.sexe[d$sexe == "Femme" & d$age >= 40] <- "Femme plus de 40 ans"
R> table(d$age.sexe)
Femme moins de 40 ans Femme plus de 40 ans Homme moins de 40 ans
376 725 315
Homme plus de 40 ans
584
Les combinaisons de variables un peu complexes nécessitent parfois un petit travail de réflexion. En particulier, l'ordre des commandes de recodage a parfois une influence dans le résultat final.
Variables scores▲
Une variable score est une variable calculée en additionnant des poids accordés aux modalités d'une série de variables qualitatives.
Pour prendre un exemple tout à fait arbitraire, imaginons que nous souhaitons calculer un score d'activités extérieures. Dans ce score on considère que le fait d'aller au cinéma « pèse » 10, celui de pêcher ou chasser vaut 30 et celui de faire du sport vaut 20. On pourrait alors calculer notre score de la manière suivante :
2.
3.
4.
5.
6.
7.
R> d$score.ext <- 0
R> d$score.ext[d$cinema == "Oui"] <- d$score.ext[d$cinema == "Oui"] + 10
R> d$score.ext[d$peche.chasse == "Oui"] <- d$score.ext[d$peche.chasse == "Oui"] + 30
R> d$score.ext[d$sport == "Oui"] <- d$score.ext[d$sport == "Oui"] + 20
R> table(d$score.ext)
0 10 20 30 40 50 60
800 342 229 509 31 41 48
Cette notation étant un peu lourde, on peut l'alléger un peu en utilisant la fonction ifelse. Celle-ci prend en argument une condition et deux valeurs. Si la condition est vraie, elle retourne la première valeur, sinon elle retourne la seconde.
2.
3.
4.
5.
6.
R> d$score.ext <- 0
R> d$score.ext <- ifelse(d$cinema == "Oui", 10, 0) + ifelse(d$peche.chasse ==
+ "Oui", 30, 0) + ifelse(d$sport == "Oui", 20, 0)
R> table(d$score.ext)
0 10 20 30 40 50 60
800 342 229 509 31 41 48
Vérification des recodages▲
Il est très important de vérifier, notamment après les recodages les plus complexes, que l'on a bien obtenu le résultat escompté. Les deux points les plus sensibles étant les valeurs manquantes et les erreurs dans les conditions.
Pour vérifier tout cela, le plus simple est sans doute de faire des tableaux croisés entre la variable recodée et celles ayant servi au recodage, à l'aide de la fonction table, et de vérifier le nombre de valeurs manquantes dans la variable recodée avec summary, freq ou table.
Par exemple :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
R> d$act.manuelles <- NA
R> d$act.manuelles[d$cuisine == "Oui" & d$bricol == "Oui"] <- "Cuisine et Bricolage"
R> d$act.manuelles[d$cuisine == "Oui" & d$bricol == "Non"] <- "Cuisine seulement"
R> d$act.manuelles[d$cuisine == "Non" & d$bricol == "Oui"] <- "Bricolage seulement"
R> d$act.manuelles[d$cuisine == "Non" & d$bricol == "Non"] <- "Ni cuisine ni bricolage"
R> table(d$act.manuelles, d$cuisine)
Non Oui
Bricolage seulement 437 0
Cuisine et Bricolage 0 416
Cuisine seulement 0 465
Ni cuisine ni bricolage 682 0
R> table(d$act.manuelles, d$bricol)
Non Oui
Bricolage seulement 0 437
Cuisine et Bricolage 0 416
Cuisine seulement 465 0
Ni cuisine ni bricolage 682 0
Tri de tables▲
On a déjà évoqué l'existence de la fonction sort, qui permet de trier les éléments d'un vecteur.
R> sort(c(2, 5, 6, 1, 8))
[1] 1 2 5 6 8On peut appliquer cette fonction à une variable, mais celle-ci ne permet que d'ordonner les valeurs de cette variable, et pas l'ensemble du tableau de données dont elle fait partie. Pour cela nous avons besoin d'une autre fonction, nommée order. Celle-ci ne renvoie pas les valeurs du vecteur triées, mais les emplacements de ces valeurs.
Un exemple pour comprendre :
R> order(c(15, 20, 10))
[1] 3 1 2Le résultat renvoyé signifie que la plus petite valeur est la valeur située en 3e position, suivie de celle en 1re position et de celle en 2e position. Tout cela ne paraît pas passionnant à première vue, mais si on mélange ce résultat avec un peu d'indexation directe, ça devient intéressant…
2.
3.
4.
5.
6.
7.
R> order(d$age)
[1] 162 215 346 377 511 646 852 916 1211 1213 1261 1333 1395 1447
[15] 1600 1774 1937 38 100 134 196 204 256 257 349 395 407 427
[29] 453 578 726 969 1052 1056 1077 1177 1234 1250 1342 1377 1381 1382
[43] 1540 1559 1607 1634 1689 1983 9 18 25 231 335 347 358 488
[57] 496 642 826 922 1023 1042 1156 1175 1290 1384 1464 1467 1608 1661
[ reached getOption("max.print") -- omitted 1930 entries ]]
Ce que cette fonction renvoie, c'est l'ordre dans lequel on doit placer les éléments de age, et donc par extension les lignes de d, pour que la variable soit triée par ordre croissant. Par conséquent, si on fait :
R> d.tri <- d[order(d$age), ]alors on a trié les lignes de d par ordre d'âge croissant ! Et si on fait un petit :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
R> head(d.tri, 3)
id age sexe nivetud poids occup qualif freres.soeurs
162 162 18 Homme <NA> 4982.964 Etudiant, eleve <NA> 2
215 215 18 Homme <NA> 4631.188 Etudiant, eleve <NA> 2
clso relig trav.imp trav.satisf hard.rock
162 Non Appartenance sans pratique <NA> <NA> Non
215 Oui Ni croyance ni appartenance <NA> <NA> Non
lecture.bd peche.chasse cuisine bricol cinema sport heures.tv fs.fac
162 Non Non Non Non Non Oui 3 2
215 Non Non Oui Non Oui Oui 2 2
fs.char qualif.char fs.num age5cl age20 age6cl qualif.reg
162 2 <NA> 2 (17.9,33.8] <20ans [18,30) <NA>
215 2 <NA> 2 (17.9,33.8] <20ans [18,30) <NA>
trav.satisf.reg ecart.heures.tv act.manuelles
162 Valeur manquante 0.7534336 Ni cuisine ni bricolage
215 Valeur manquante -0.2465664 Cuisine seulement
age.sexe score.ext
162 Homme moins de 40 ans 20
215 Homme moins de 40 ans 30
[getOption("max.print") est atteint -- dernière ligne omises ]]
on a les caractéristiques des trois enquêtés les plus jeunes.
On peut évidemment trier par ordre décroissant en utilisant l'option decreasing=TRUE. On peut donc afficher les caractéristiques des trois individus les plus âgés avec :
R> head(d[order(d$age, decreasing = TRUE), ], 3)Fusion de tables▲
Lorsque l'on traite de grosses enquêtes, notamment les enquêtes de l'INSEE, on a souvent à gérer des données réparties dans plusieurs tables, soit du fait de la construction du questionnaire, soit du fait de contraintes techniques (fichiers dbf ou Excel limités à 256 colonnes, par exemple).
Une opération relativement courante consiste à fusionner plusieurs tables pour regrouper tout ou partie des données dans un unique tableau.
Nous allons simuler artificiellement une telle situation en créant deux tables à partir de l'extrait de l'enquête Histoire de vie :
2.
3.
4.
5.
6.
7.
8.
9.
10.
R> data(hdv2003)
R> d <- hdv2003
R> dim(d)
[1] 2000 20
R> d1 <- subset(d, select = c("id", "age", "sexe"))
R> dim(d1)
[1] 2000 3
R> d2 <- subset(d, select = c("id", "clso"))
R> dim(d2)
[1] 2000 2
On a donc deux tableaux de données, d1 et d2, comportant chacun 2000 lignes et respectivement 3 et 2 colonnes. Comment les rassembler pour n'en former qu'un ?
Intuitivement, cela paraît simple. Il suffit de « coller » d2 à la droite de d1, comme dans l'exemple suivant.

Cela semble fonctionner. La fonction qui permet d'effectuer cette opération sous R s'appelle cbind, elle « colle » des tableaux côte à côte en regroupant leurs colonnes(18).
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
R> cbind(d1, d2)
id age sexe id clso
1 1 28 Femme 1 Oui
2 2 23 Femme 2 Oui
3 3 59 Homme 3 Non
4 4 34 Homme 4 Non
5 5 71 Femme 5 Oui
6 6 35 Femme 6 Non
7 7 60 Femme 7 Oui
8 8 47 Homme 8 Non
9 9 20 Femme 9 Oui
10 10 28 Homme 10 Non
11 11 65 Femme 11 Oui
12 12 47 Homme 12 Oui
13 13 63 Femme 13 Oui
14 14 67 Femme 14 Oui
[getOption("max.print") est atteint -- 1986 lignes omises ]]
À part le fait que l'on a une colonne id en double, le résultat semble satisfaisant. À première vue seulement. Imaginons maintenant que nous avons travaillé sur d1 et d2, et que nous avons ordonné les lignes de d1 selon l'âge des enquêtés :
R> d1 <- d1[order(d1$age), ]Répétons l'opération de collage :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
R> cbind(d1, d2)
id age sexe id clso
162 162 18 Homme 1 Oui
215 215 18 Homme 2 Oui
346 346 18 Femme 3 Non
377 377 18 Homme 4 Non
511 511 18 Homme 5 Oui
646 646 18 Homme 6 Non
852 852 18 Femme 7 Oui
916 916 18 Femme 8 Non
1211 1211 18 Homme 9 Oui
1213 1213 18 Femme 10 Non
1261 1261 18 Homme 11 Oui
1333 1333 18 Femme 12 Oui
1395 1395 18 Homme 13 Oui
1447 1447 18 Femme 14 Oui
[getOption("max.print") est atteint -- 1986 lignes omises ]]
Que constate-t-on ? La présence de la variable id en double nous permet de voir que les identifiants ne coïncident plus ! En regroupant nos colonnes, nous avons donc attribué à des individus les réponses d'autres individus.
La commande cbind ne peut en effet fonctionner que si les deux tableaux ont exactement le même nombre de lignes, et dans le même ordre, ce qui n'est pas le cas ici.
On va donc être obligé de procéder à une fusion des deux tableaux, qui va permettre de rendre à chaque ligne ce qui lui appartient. Pour cela nous avons besoin d'un identifiant qui permet d'identifier chaque ligne de manière unique et qui doit être présent dans tous les tableaux. Dans notre cas, c'est plutôt rapide, il s'agit de la variable id.
Une fois l'identifiant reconnu(19), on peut utiliser la commande merge. Celle-ci va fusionner les deux tableaux en supprimant les colonnes en double et en regroupant les lignes selon leurs identifiants :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
R> d.complet <- merge(d1, d2, by = "id")
R> d.complet
id age sexe clso
1 1 28 Femme Oui
2 2 23 Femme Oui
3 3 59 Homme Non
4 4 34 Homme Non
5 5 71 Femme Oui
6 6 35 Femme Non
7 7 60 Femme Oui
8 8 47 Homme Non
9 9 20 Femme Oui
10 10 28 Homme Non
11 11 65 Femme Oui
12 12 47 Homme Oui
13 13 63 Femme Oui
14 14 67 Femme Oui
15 15 76 Femme Oui
16 16 49 Femme Non
17 17 62 Homme Non
[getOption("max.print") est atteint -- 1983 lignes omises ]]
Ici l'utilisation de la fonction est plutôt simple, car nous sommes dans le cas de figure idéal : les lignes correspondent parfaitement et l'identifiant est clairement reconnu. Parfois les choses peuvent être un peu plus compliquées :
- parfois les identifiants n'ont pas le même nom dans les deux tableaux. On peut alors les spécifier par les options by.x et by.y ;
- parfois les deux tableaux comportent des colonnes (hors identifiants) ayant le même nom. merge conserve dans ce cas ces deux colonnes, mais les renomme en les suffixant par .x, pour celles provenant du premier tableau et .y pour celles du second ;
- parfois on n'a pas d'identifiant unique préétabli, mais on en construit un à partir de plusieurs variables. On peut alors donner un vecteur en paramètres de l'option by, par exemple
by=c("nom","prenom","date.naissance").
Une subtilité supplémentaire intervient lorsque les deux tableaux fusionnés n'ont pas exactement les mêmes lignes. Par défaut, merge ne conserve que les lignes présentes dans les deux tableaux :
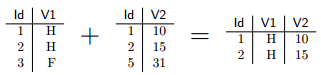
On peut cependant modifier ce comportement avec les options all.x=TRUE et all.y=TRUE. La première option indique de conserver toutes les lignes du premier tableau. Dans ce cas merge donne une valeur NA pour ces lignes aux colonnes provenant du second tableau. Ce qui donnerait :
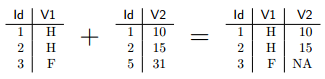
all.y fait la même chose en conservant toutes les lignes du second tableau. On peut enfin décider de conserver toutes les lignes des deux tableaux en utilisant à la fois all.x=TRUE et all.y=TRUE, ce qui donne :
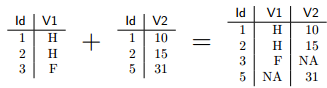
Parfois, l'un des identifiants est présent à plusieurs reprises dans l'un des tableaux (par exemple, lorsque l'une des tables est un ensemble de ménages et que l'autre décrit l'ensemble des individus de ces ménages). Dans ce cas les lignes de l'autre table sont dupliquées autant de fois que nécessaire :
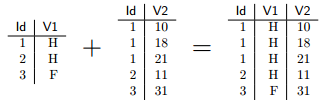
Organiser ses scripts▲
Il ne s'agit pas ici de manipulation de données à proprement parler, mais plutôt d'une conséquence de ce qui a été vu précédemment : à mesure que recodages et traitements divers s'accumulent, votre script R risque de devenir rapidement très long et pas très pratique à éditer.
Il est très courant de répartir son travail entre différents fichiers, ce qui est rendu très simple par la fonction source. Celle-ci permet de lire le contenu d'un fichier de script et d'exécuter son contenu.
Prenons tout de suite un exemple. La plupart des scripts R commencent par charger les extensions utiles, par définir le répertoire de travail à l'aide de setwd, à importer les données, à effectuer manipulations, traitements et recodages, puis à mettre en œuvre les analyses. Prenons le fichier fictif suivant :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
23.
24.
library(rgrs)
library(foreign)
setwd("/home/julien/r/projet")
## IMPORT DES DONNÉES
d1 <- read.dbf("tab1.dbf")
d2 <- read.dbf("tab2.dbf")
d <- merge(d1, d2, by="id")
## RECODAGES
d$tx.chomage <- as.numeric(d$tx.chomage)
d$pcs[d$pcs == "Ouvrier qualifie"] <- "Ouvrier"
d$pcs[d$pcs == "Ouvrier specialise"] <- "Ouvrier"
d$age5cl <- cut(d$age, 5)
## ANALYSES
tab <- table(d$tx.chomage, d$age5cl)
tab
chisq.test(tab)
Une manière d'organiser notre script(20) pourrait être de placer les opérations d'import des données et celles de recodage dans deux fichiers scripts séparés. Créons alors un fichier nommé import.R dans notre répertoire de travail et copions les lignes suivantes :
2.
3.
4.
5.
6.
## IMPORT DES DONNÉES
d1 <- read.dbf("tab1.dbf")
d2 <- read.dbf("tab2.dbf")
d <- merge(d1, d2, by="id")
Créons également un fichier recodages.R avec le contenu suivant :
2.
3.
4.
5.
6.
7.
## RECODAGES
d$tx.chomage <- as.numeric(d$tx.chomage)
d$pcs[d$pcs == "Ouvrier qualifie"] <- "Ouvrier"
d$pcs[d$pcs == "Ouvrier specialise"] <- "Ouvrier"
d$age5cl <- cut(d$age, 5)
Dès lors, si nous rajoutons les appels à la fonction source qui vont bien, le fichier suivant sera strictement équivalent à notre fichier de départ :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
library(rgrs)
library(foreign)
setwd("/home/julien/r/projet")
source("import.R")
source("recodages.R")
## ANALYSES
tab <- table(d$tx.chomage, d$age5cl)
tab
chisq.test(tab)
Au fur et à mesure du travail sur les données, on placera les recodages que l'on souhaite conserver dans le fichier recodages.R.
Cette méthode présente plusieurs avantages :
- bien souvent, lorsque l'on effectue des recodages on se retrouve avec des variables recodées que l'on ne souhaite pas conserver. Si on prend l'habitude de placer les recodages intéressants dans le fichier recodages.R, alors il suffit d'exécuter les cinq premières lignes du fichier pour se retrouver avec un tableau de données d propre et complet ;
- on peut répartir ses analyses dans différents scripts. Il suffit alors de copier les cinq premières lignes du fichier précédent dans chacun des scripts, et on aura l'assurance de travailler sur exactement les mêmes données.
Le premier point illustre l'une des caractéristiques de R : il est rare que l'on stocke les données modifiées. En général on repart toujours du fichier source original, et les recodages sont conservés sous forme de scripts et recalculés à chaque fois que l'on recommence à travailler. Ceci offre une traçabilité parfaite du traitement effectué sur les données.
Exercices▲
? Solution
Renommer la variable clso du jeu de données hdv2003 en classes.sociales, puis la renommer en clso.
? Solution
Réordonner les niveaux du facteur clso pour que son tri à plat s'affiche de la manière suivante :
tmp
Non Ne sait pas Oui
1037 27 936? Solution
Affichez :
- les 3 premiers éléments de la variable cinema ;
- les éléments 12 à 30 de la variable lecture.bd ;
- les colonnes 4 et 8 des lignes 5 et 12 du jeu de données hdv2003 ;
- les 4 derniers éléments de la variable age.
? Solution
Construisez les sous-tableaux suivants avec la fonction subset :
- âge et sexe des lecteurs de BD ;
- ensemble des personnes n'étant pas chômeur (variable occup), sans la variable cinema ;
- identifiants des personnes de plus de 45 ans écoutant du hard rock ;
- femmes entre 25 et 40 ans n'ayant pas fait de sport dans les douze derniers mois ;
- hommes ayant entre 2 et 4 frères et sœurs et faisant la cuisine ou du bricolage.
? Solution
Calculez le nombre moyen d'heures passées devant la télévision chez les lecteurs de BD, d'abord en construisant les sous-populations, puis avec la fonction tapply.
? Solution
Convertissez la variable freres.soeurs en variable de type caractères. Convertissez cette nouvelle variable en facteur. Puis convertissez à nouveau ce facteur en variable numérique. Vérifiez que votre variable finale est identique à la variable de départ.
? Solution
Découpez la variable freres.soeurs :
- en cinq classes d'amplitude égale ;
- en catégories « de 0 à 2 », « de 3 à 4 », « plus de 4 », avec les étiquettes correspondantes ;
- en quatre classes d'effectif équivalent ;
- d'où vient la différence d'effectifs entre les deux découpages précédents ?
? Solution
Recodez la variable trav.imp en trav.imp2cl pour obtenir les modalités « Le plus ou aussi important » et « moins ou peu important ». Vérifiez avec des tris à plat et un tableau croisé.
Recodez la variable relig en relig.4cl en regroupant les modalités « Pratiquant regulier » et « Pratiquant occasionnel » en une seule modalité « Pratiquant », et en remplaçant la modalité « NSP ou NVPR » par des valeurs manquantes. Vérifiez avec un tri croisé.
Créez une variable ayant les modalités suivantes :
- homme de plus de 40 ans lecteur de BD ;
- homme de plus de 30 ans ;
- femme faisant du bricolage ;
- autre.
Vérifier avec des tris croisés.
? Solution
Ordonner le tableau de données selon le nombre de frères et sœurs croissant. Afficher le sexe des 10 individus regardant le plus la télévision.
 R pour les sociologues (et assimilés)
R pour les sociologues (et assimilés)


